RNA polimerasi
Quale ruolo svolge la RNA polimerasi?
Il termine RNA Polimerasi identifica una famiglia di enzimi complessi, costituiti da numerose sub-unità proteiche, che consentono la sintesi di un filamento di RNA complementare ad uno dei due filamenti di DNA che compongono la doppia elica.
Il processo che si determina, noto come trascrizione del DNA, è il primo passaggio necessario perché vengano sintetizzate le proteine. Non tutti gli RNA contengono le sequenze necessarie alla sintesi proteica; queste sono contenute solo nei cosiddetti RNA messaggeri (mRNA).
Oltre a questi, l'RNA-Polimerasi sintetizza anche filamenti di RNA con funzione di trasporto degli amminoacidi (t-RNA) e costituenti dei ribosomi (r-RNA). Altre piccole molecole di RNA hanno funzioni che si svolgono nel nucleo (snRNA).
Inoltre esistono alcune molecole di RNA che hanno funzione enzimatica o strutturale al pari delle molecole proteiche. Nei procarioti esiste un unico enzima che catalizza la sintesi di ognuno di questi tipi di RNA, mentre negli eucarioti si identificano tre enzimi differenti, ognuno deputato alla sintesi di alcune delle classi di RNA menzionate in precedenza.
Struttura e funzionamento generale dell'RNA-Polimerasi
L'RNA-Polimerasi appartiene al gruppo dei cosiddetti oloenzimi, enzimi cioè costituiti da più sub-unità tutte necessarie allo svolgimento delle proprie funzioni.
In linea generale, le due sub-unità maggiori presentano i siti attivi ove avviene la catalisi della reazione di formazione del legame fosfodiesterico; uno (nei procarioti) o più di un (negli eucarioti) cofattore sono necessari per il riconoscimento delle sequenze promotore all'interno del gene e l'avvio della trascrizione.
La reazione prevede l'attacco idrofilico del gruppo ossidrile (OH) in posizione 3' del nucleotide già presente al gruppo fosfato del nucleotide successivo, con rilascio di una molecola di Pirofosfato.
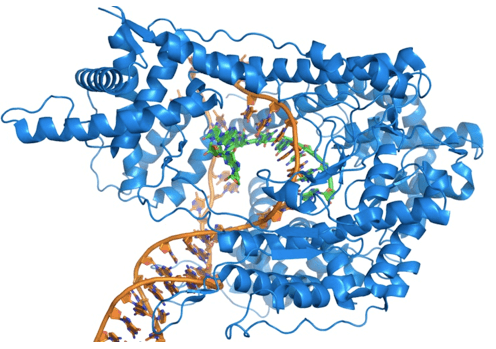
Figura 1: RNA Polimerasi in funzione. In Blu l'oloenzima; in arancio le doppia elica del DNA; In Verde il filamento di RNA sintetizzato.
Il processo trascrizionale mediato dall'azione della RNA-Polimerasi avviene unicamente in direzione 5'-3' e coinvolge un solo filamento della doppia elica, detto filamento stampo.
Il prodotto della trascrizione, ossia il filamento di RNA, sarà complementare allo stampo e uguale all'altro filamento, definito filamento codificante, con eccezione della presenza di Uracile al posto della Timina e dell'utilizzo di zucchero Ribosio al posto del Desossiribosio.
L'RNA -Polimerasi ha anche funzione di proofreading, ossia di correzione di bozze. Tale funzione si esercita attraverso due vie:
- editing pirofosfolitico: il nucleotide inserito in maniera errata è rimosso mediante reincorporazione di due gruppi fosfato nel sito attivo dell'enzima
- editing idrolitico: l'enzima torna indietro di alcune basi, taglia un piccolo pezzo di RNA che contiene il nucleotide sbagliato e ricomincia la sua azione
Tutte le RNA Polimerasi svolgono la propria attività di trascrizione del DNA seguendo tre fasi: Inizio, Allungamento del filamento e Terminazione. Il legame dei cofattori è determinante per l'avvio del procedimento, la sua rimozione è invece fondamentale per il processo di allungamento.
Le reazioni di incorporazione dei nucleotidi successivi richiedono l'utilizzo di ioni metallici, precisamente di cationi Mg2+, e l'enzima è in grado di iniziare la sintesi di un filamento senza bisogno di uno starter, come invece avviene per la DNA- Polimerasi.
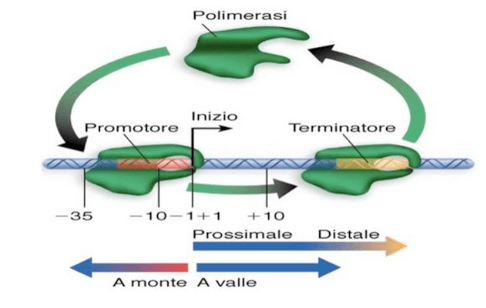
Figura 2: L'insieme dei siti Promotore e Terminatore e la sequenza codificante definisce la cosiddetta "Unità Trascrizionale".
RNA-Polimerasi Batterica: struttura e dettagli di funzionamento
La RNA-Polimerasi batterica è stata oggetto di approfonditi studi soprattutto in Escherichia coli, uno dei batteri in assoluto meglio conosciuti.
In esso, la RNA Polimerasi è composta da 4 sub-unità più il fattore sigma (σ). Il centro catalitico è formato dalle due sub-unità maggiori, β e β' (grandi rispettivamente 155 e 160 kD); due sub-unità identiche, dette sub-unità ɑ (40 kD) sono responsabili dell'assemblaggio dell'enzima e del legame con eventuali attivatori. Il fattore σ, presente solo all'inizio della trascrizione, è invece determinante per il riconoscimento delle sequenze del promotore.
Le sub-unità maggiori formano una struttura definita a "chela di granchio", in cui le estremità della chela si avvolgono intorno al DNA mentre alla base delle chele si trova il sito attivo dove si legano 2 ioni Mg2+ e dove si crea il solco centrale attivo. La RNA- Polimerasi lega in maniera non selettiva il DNA in un punto casuale.
In questa fase il DNA è a doppia elica. Il legame con il fattore σ determina la capacità di riconoscere il promotore e dare inizio alla trascrizione. Il legame con il sito -35 del promotore dà origine al cosiddetto "complesso chiuso" formato da RNA- Polimerasi e DNA. Il legame con il sito -10 determina una isomerizzazione dell'oloenzima senza consumo di ATP e l'apertura della doppia elica del DNA (formazione del "complesso aperto") con conseguente inizio della trascrizione.
L'azione del fattore σ è determinante perché riduce l'affinità dell'RNA Polimerasi per le sequenze di DNA diverse da quelle del promotore permettendone il riconoscimento. Dopo una fase definita "inizio abortivo" (in cui vengono prodotti solo circa 10 nucleotidi con una certa difficoltà) il fattore σ si stacca, l'oloenzima acquista maggior affinità per il DNA e si ha la fase di allungamento. In corrispondenza delle sequenze di interruzione si ha la fine del processo. I terminatori possono possedere capacità intrinseche di interruzione del processo o necessitare dell'azione di proteine di supporto, dette proteine RHO (terminatori RHO-Dipendenti).
Il passaggio del DNA, dei nucleotidi in entrata e del filamento di RNA avviene attraverso 5 canali: il canale NTP-Uptake permette l'ingresso dei nucleotidi da incorporare; l'RNA-Exit guida l'uscita del filamento di RNA sintetizzato; il DNA- downstream incanala il filamento di DNA a doppia elica che viene quindi aperto. I due filamenti seguono due percorsi differenti: il filamento stampo segue il canale Template mentre il filamento codificante segue il canale Non Template. I due filamenti si riuniscono poi a monte dell'enzima.
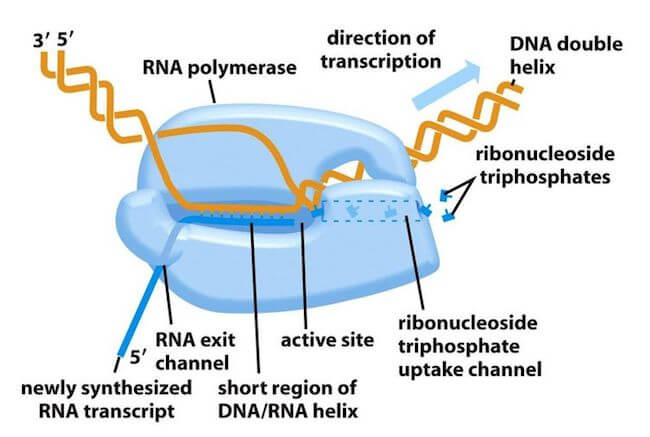
Figura 3: RNA Polimerasi Batterica.
Altre RNA Polimerasi
Negli eucarioti le DNA Polimerasi sono tre, il cui funzionamento segue a grandi linee quello delle RNA procariotiche.
La RNA-Polimerasi I sintetizza gli rRNA; la II sintetizza gli mRNA e alcuni RNA nucleari; la III sintetizza i tRNA, gli rRNA 5s e un solo tipo di RNA nucleare.
Nei virus è presente infine un ulteriore tipo di enzima coinvolto nella replicazione del genoma dei virus a RNA, detta RNA Polimerasi RNA-Dipendente.
Link correlati:
Quanto è lungo il DNA?
Quali sono gli acidi nucleici?
Cosa sono i fattori di trascrizione?
Funzioni e struttura dell'apparato di Golgi
Che cos'è l'acido ribonucleico (RNA)?
Che cos'è la reazione a catena della polimerasi o PCR?
Studia con noi