Reazione a catena della polimerasi (PCR)
Che cos'è la reazione a catena della polimerasi o PCR?
PCR è l'acronimo di reazione a catena della polimerasi, dall'inglese "polymerase chain reaction" ed indica una tecnica che permette di amplificare gli acidi nucleici.
Amplificare significa ottenere numerose copie del frammento di acido nucleico di interesse, si tratta di una tecnica necessaria poiché per molte metodiche di biologia molecolare importanti nelle biotecnologie, il quantitativo di acidi nucleici, di DNA per esempio, estratto da un campione di cellule o tessuto non è sufficiente per le analisi da condurre (ad esempio il sequenziamento, oppure l'analisi dei micro satelliti).
Ricordiamo che di ogni gene, sono presenti due sole copie in ogni cellula umana, due alleli che possono essere differenti tra loro.
Prima dell'invenzione della tecnica della PCR, nel 1983, i biologi utilizzavano il clonaggio per amplificare frammenti di DNA di loro interesse.
Si tratta di una tecnica molto laboriosa, resa possibile dalla scoperta degli enzimi di restrizione: enzimi che sono in grado di riconoscere brevi sequenze di nucleotidi, spesso palindrome, e di operare un taglio netto dei due filamenti oppure un taglio che crea estremità coesive.
Il DNA di interesse viene tagliato con un enzima di restrizione noto e lo stesso avviene per un adeguato vettore di clonaggio (elemento di DNA che possieda sequenze per la duplicazione in cellule competenti), quindi il frammento è inserito nel vettore e tramite una ligasi si procede al legame tra essi.
Il vettore di clonaggio, inserito in cellule competenti (adeguate, ad esempio batteriche se si tratta di un vettore batterico), viene duplicato più e più volte, duplicando anche il frammento di DNA di interesse che dovrà poi essere selezionato, estratto, tagliato dal vettore stesso e sarà quindi, solo allora, possibile procedere con l'analisi.
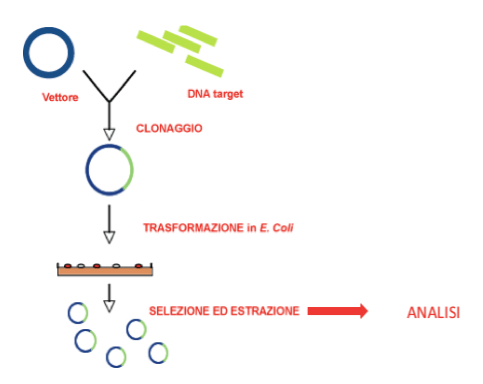
Schema della tecnica di clonaggio per amplificare un frammento di DNA di interesse.
L'inventore di questa tecnica è Kary Mullis (Lenoir, 28 dicembre 1944 – Newport Beach, 7 agosto 2019), biochimico statunitense decisamente eccentrico e controverso per alcune sue posizioni ad esempio sul virus dell'HIV. L'invenzione della PCR risale al 1983, per la quale, Mullis è stato insignito del premio Nobel nel 1993.
Si tratta, dunque, di una relativamente recente acquisizione della biologia molecolare e dunque delle biotecnologie, ma certamente di fondamentale importanza, forse proprio al pari della scoperta degli enzimi di restrizione, per l'avanzamento delle biotecnologie stesse.
La tecnica della PCR si basa su alcune caratteristiche del DNA:
- la doppia elica può essere denaturata e rinaturata sia intervenendo sulla concentrazione salina del tampone in cui è posta, sia sulla temperatura. Quest'ultima è senza dubbio più immediata e semplice da modificare in laboratorio: l'innalzamento della temperatura fornisce l'energia che consente la rottura dei ponti a idrogeno tra le basi azotate appaiate per complementarietà, l'abbassamento della temperatura induce, invece, la formazione di nuovi ponti a idrogeno secondo le regole della complementarietà. Va notato che i filamenti di acidi nucleici (DNA o RNA) si riappaiano tra loro solo sulla base della complementarietà delle basi azotate dei nucleotidi, dunque si possono anche formare degli etero duplex tra un filamento di DNA ed uno di RNA;
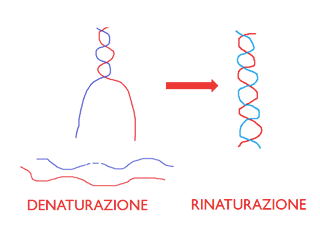
Denaturazione e rinaturazione della doppia elica.
- la polimerizzazione degli acidi nucleici, nella cellula, prevede l'utilizzo di nucleotidi trifosfati e dell'enzima polimerasi, in particolare per la duplicazione del DNA l'enzima chiave è la DNA polimerasi DNA dipendente che è in grado di catalizzare il legame fosfodiestere tra il 5' fosforilato di un nuovo nucleotide e l'ossidrile in 3' del nucleotide precedente, utilizzando l'energia della idrolisi del pirofosfato dal nucleotide trifosfato.
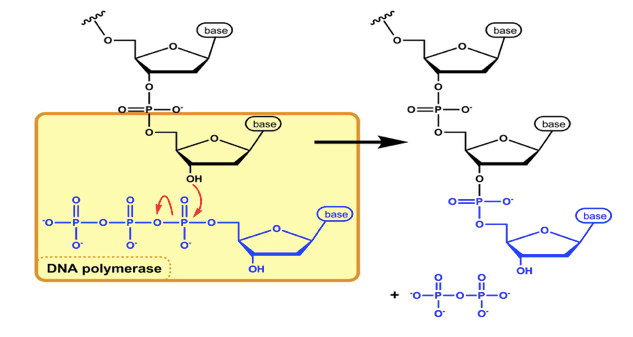
Formazione del legame sfosfodiestere, direzione di sintesi 5'→3'.
In vitro è stato possibile ripetere le tappe principali della duplicazione del DNA che avviene nelle cellule grazie alla possibilità, come già detto, di denaturare la doppia elica quindi permettere l'accesso ai nucleotidi che devono fungere da stampo ed alla scoperta della polimerasi del batterio Thermus acquaticus.
Si tratta di un batterio termofilo, isolato per la prima volta nelle pozze di acqua calda del parco nazionale di Yellowstone, negli USA, un ambiente estremo con temperature elevate.
Questo batterio possiede, quindi, enzimi in grado di resistere ad alte temperature, in particolare la sua polimerasi è stabile (non si denatura) a 95° C e presenta come temperatura ottimale di catalisi 72° C.
Oggi la polimerasi del batterio termofilo è facilmente reperibile commercialmente ed è prodotta, non più in maniera estrattiva, ma con la tecnica del DNA ricombinante.
La miscela di reazione in vitro contiene un buffer salino, la Taq (abbreviazione di DNA polimerasi del batterio termofilo), i dNTP ovvero una miscela di desossinucleotidi trifosfati, il DNA di interesse che deve fungere da stampo per la sua amplificazione e una coppia di primers.
Con il termine primer si indica un oligonucleotide (RNA) di innesco di cui necessita la DNA polimerasi (e dunque la Taq) per cominciare la reazione di polimerizzazione.
In vitro i primers utilizzati sono due e devono essere disegnati fiancheggianti al DNA templato di interesse (esistono software specifici per il loro disegno anche se spesso i ricercatori li cercano in letteratura).
Sono indicati con R ed F (foward e reverse) poiché devono appaiarsi rispettivamente al filamento veloce e a quello lento, sono costituiti da ribonucleotidi e caratterizzati da una specifica temperatura di meltig (quella alla quale il primer è associato al templato in maniera specifica).
Nel disegnare i primers occorre evitare sequenze complementari all'interno della coppia per evitare che essi si leghino uno all'altro e non al templato, rispettare la lunghezza massima di 20-22 paia di basi, evitare sequenze palindrome che causerebbero ripiegamenti.
Le fasi della PCR
LA PCR in laboratorio è effettuata grazie ai reagenti di cui abbiamo già discusso e al termociclatore: uno strumento in grado di variare la temperatura del blocco nel quale vengono alloggiate le provette o le piastre, in maniera sufficientemente veloce da consentire la reazione di amplificazione in tempi decisamente brevi, nell'ordine dell'ora.

Esempio di termociclatore disponibile in commercio.
La reazione a catena della polimerasi prevede le seguenti fasi:
1) denaturazione: la prima fase prevede di portare la miscela di reazione a 95° C per 3,5 minuti al fine di denaturare tutto l'acido nucleico presente (si potrebbe trattare di DNA genomico estratto da cellule in coltura o da tessuti ma anche di DNA clonato con il metodo descritto in precedenza oppure derivante da altre tecniche di biologia molecolare);
2) seguono tre fasi che mediamente durano ciascuna dai 30 ai 90 secondi, che vengono ripetute per circa 35-40 volte:
- denaturazione: a 95° C che consente il riottenimento di due filamenti separati dopo ogni ciclo di amplificazione;
- annealing: è la fase in cui i primers riconoscono e legano il templato; avviene alla temperatura alla quale i primers risultano legati per complementarietà al templato, dipende dalla temperatura di melting degli stessi ed è ottenuta sperimentalmente per ogni coppia di primers;
- elongation: allungamento del filamento, avviene a 72° C, si tratta della vera e propria fase di polimerizzazione da parte della Taq.
3) si ha infine una ultima fase che dura alcuni minuti (5-10) di allungamento finale a 72° C che consente alla polimerasi di completare le eventuali catene incomplete, quindi la miscela di reazione è portata a 4° C: temperatura alla quale il DNA si conserva per diverso tempo.
Il susseguirsi di questi cicli di amplificazione consente di ottenere molte copie del DNA di interesse a partire dalle iniziali; ad ogni ciclo le copie di DNA vengono duplicate secondo lo schema rappresentato di seguito:
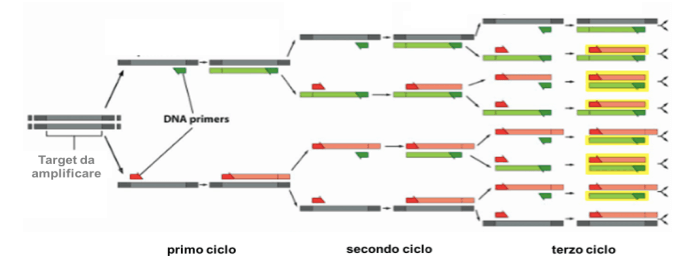
Schema dell'amplificazione esponenziale del templato nel corso delle PCR.
Si ottiene un'amplificazione esponenziale del templato secondo la resa teorica riportata di seguito; in verità la resa reale sarà un po' più bassa poiché l'enzima non lavora con un'efficienza del 100% e i reagenti man mano che la reazione procede, diminuiscono la loro concentrazione nella miscela di reazione, contribuendo ad abbassare l'efficienza:
Resa teoria = 2n · y
in cui:
- y = numero iniziale di copie di DNA;
- n = numero di cicli di PCR.
Secondo questa formula, partendo, ad esempio, da 100 copie del templato di interesse, si ottengono, dopo 30 cicli di amplificazione, molecole nell'ordine di 1012 ovvero delle migliaia di miliardi.
Link correlati:
Le cellule nervose: il neurone
Che cosa sono i trasposoni?
Che cosa è l'ereditarietà?
Che cos'è il il ciclo di Cori?
Studia con noi