Riparazione del DNA
Come avviene la riparazione del DNA?
Il DNA è la molecola organica più importante, che custodisce al proprio interno le istruzioni per la costruzione e il funzionamento di tutte le cellule.
Un errore nella sequenza di nucleotidi determina un errore nell'informazione contenuta e quindi un rischio per la sopravvivenza. Per questo motivo le cellule possiedono sofisticati sistemi di riparazione dei danni del DNA, in grado di riconoscere e rimediare ad eventuali errori. Ciononostante, non sempre gli errori vengono riconosciuti e riparati.
Alla base dell'evoluzione, infatti, c'è propria la possibilità che nuove combinazioni del codice genetico emergano attraverso errori che, non riparati, si fissano nel DNA e danno luogo a mutazioni.
Riparazione del DNA: errori e danni
La necessità di riparazione del DNA può nascere in conseguenza di un errore durante la replicazione oppure di un danneggiamento a carico del DNA al di fuori della replicazione.
Durante la replicazione possono verificarsi errori semplici, in cui una base azotata è sostituita con un'altra, o errori complessi, in cui una o più basi azotate sono sottratte o aggiunte alla sequenza, cambiando completamente l'ordine di lettura e di conseguenza modificando del tutto il codice.
Nel caso di errori semplici, i meccanismi di riparazione che possono essere esercitati sono il proofreading e il mismatch repair. Nel caso di errori complessi, non esistono sistemi di riparazione adeguati.
Oltre agli errori in fase di replicazione, possono verificarsi dei danni al DNA dovuti all'azione di agenti chimici o fenomeni fisici, che in genere danneggiano l'integrità delle basi azotate o modificano i rapporti tra di esse, causando problemi che si manifestano in occasione del primo ciclo di replicazione.
Meccanismi di riparazione del DNA: il proofreading
Durante la replicazione del DNA, la DNA Polimerasi può commettere un errore e incorporare un nucleotide sbagliato.
Quando viene incorporato un nucleotide della stessa famiglia di quello che sarebbe dovuto essere incorporato (una pirimidina è sostituita con un'altra pirimidina, oppure una purina è sostituita da un'altra purina), si parla di transizione, altrimenti si parla di transversione.
Quando questo accade, nella maggioranza dei casi la riparazione è operata grazie alla capacità di proofreading della DNA Polimerasi stessa.
Quando il filamento neosintetizzato contiene un errore, infatti, l'estremità 3' si appaierà in maniera scorretta all'altro filamento. Tale stortura tridimensionale rallenta la velocità di scorrimento della Polimerasi, diminuisce l'affinità tra il filamento neosintetizzato e il sito attivo della Polimerasi e aumenta invece l'affinità per il sito esonucleasico.
L'estremità 3', quindi, si sposta sul sito esonucleasico 3' – 5' e la Polimerasi inizia ad agire da esonucleasi, rimuovendo i nucleotidi inseriti erroneamente. Una volta eliminati gli errori, l'affinità del filamento 3' per il sito attivo della polimerasi è ripristinato, il filamento si sposta nuovamente e la polimerizzazione del nuovo DNA riprende.
Nel caso in cui l'errore sfugga all'attività di proofreading, potrà essere intercettato e risolto dal meccanismo del mismatch repair.
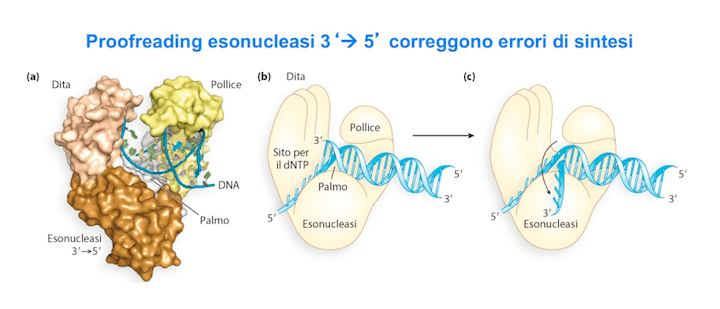
Proofreading. L'estremità 3' del filamento cambia, in caso di errore, diventa più affine al sito esonucleasico e si sposta per essere sottoposto alla rimozione dei nucleotidi sbagliati grazie alla capacità esonucleasica 3'-5' dela DNA Polimerasi.
Meccanismi di riparazione del DNA: il mismatch repair
Quello del mismatch repair è un processo che avviene durante il primo ciclo di replicazione, con modalità leggermente differenti tra procarioti ed eucarioti.
Nei procarioti l'enzima che esegue la scansione del DNA prima della replicazione è il MUT-S, un enzima dimerico che analizza la doppia elica alla ricerca di punti in cui la molecola risulta ripiegata in maniera innaturale, a causa della mancanza di corrispondenza tra le basi azotate dei due filamenti.
Nel caso in cui MUT-S rilevi un errore, si lega alla doppia elica in quel punto e recluta altri due enzimi, MUT-L e MUT-H.
MUT-H opera il taglio del filamento sbagliato, che viene riconosciuto grazie all'azione dell'enzima DAM metilasi. DAM metilasi, infatti, aggiunge un gruppo metile a tutte le adenina presenti nelle sequenze GATC.
Il filamento neosintetizzato (quindi quello errato) presenterà degli errori e risulterà metilato a metà (emi-metilato) permettendo al MUT-H di riconoscerlo e attaccarlo. Una volta che MUT-H ha legato il filamento da correggere, entrano in gioco le elicasi, le esonucleasi, la DNA Polimerasi e le ligasi che operano la riparazione.
Negli eucarioti il processo è simile ma: l'enzima MUT-S è sostituito da MSH, MUT-L è sostituito da MLH mentre MUT-H non ha analoghi.
Il riconoscimento del filamento neosintetizzato avviene attraverso il riconoscimento delle incisioni dei frammenti di Okazaki, associati alla pinza di scivolamento.
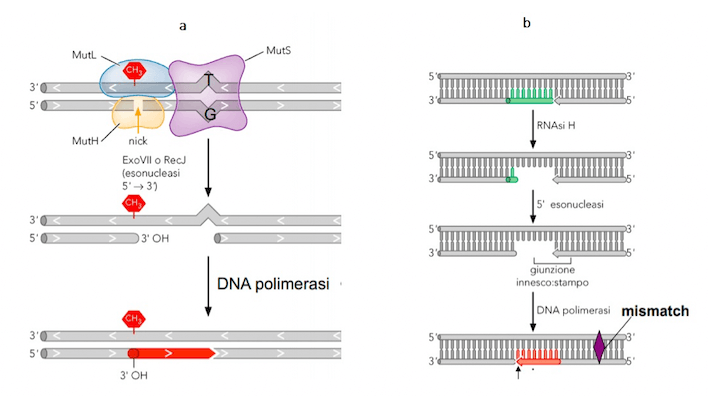
Mismatch repair nei procarioti e negli eucarioti (a,b).
Riparazione dei danni
In caso di danneggiamento dovuto all'azione di agenti chimici o fenomeni fisici (come l'esposizione ai raggi UV, l'attacco di gruppi alchilici o di specie altamente ossidanti) possono verificarsi eventi che comportano un danneggiamento dei nucleotidi.
Il caso più frequente è l'idrolisi di uno dei legami che formano i nucleotidi. Quando ad idrolizzarsi è il legame che tiene insieme la base azotata allo zucchero pentoso, la base azotata viene perduta e si parla di depurinazione.
Nel caso in cui invece avvenga l'idrolisi del gruppo amminico sulla base azotata, si parla di deaminazione.
La depurinazione, se non corretta, porta alla perdita di una coppia di basi durante la successiva replicazione; la deaminazione invece comporta in genere un mismatch, cioè un errore nella composizione del filamento neosintetizzato, perché la base deaminata si appaia con una base diversa rispetto a quella con cui si appaierebbe la base integra.
L'irradiazione, invece, cioè l'esposizione ai raggi UV, può causare danni di diversa natura. Un tipico esempio è la dimerizzazione, in cui due basi azotate poste in sequenza lungo catena di nucleotidi formano tra loro un legame covalente. In base al tipo di base che dimerizza, possono verificarsi il blocco della replicazione o la morte cellulare.
I meccanismi di riparazione di questi danni coinvolgono: l'escissione delle basi azotate sbagliate, mediante esonucleasi o glicosilasi, e la riparazione per ricombinazione (omologa o non omologa).
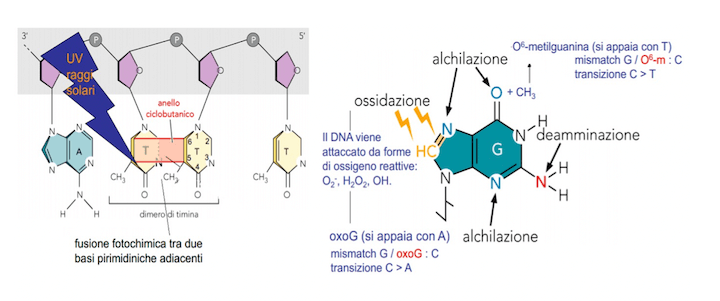
Alcuni danni al DNA dovuti all'azione di agenti chimici o fisici: dimerizzazione delle basi azotate di timina (sx) ; ossidazione, deaminazione, alchilazione (dx).
Link correlati:
Quanto è lungo il DNA?
Che cos'è il sequenziamento del DNA?
Che cos'è la reazione a catena della polimerasi o PCR?
Studia con noi