DNA polimerasi o trascrittasi inversa
Quale ruolo svolgono le DNA polimerasi o trascrittasi inversa?
L'espressione DNA polimerasi (o trascrittasi inversa) è utilizzata per individuare i componenti di una famiglia di enzimi e complessi multiproteici coinvolti a vario titolo nella sintesi e replicazione delle molecole di DNA.
Le DNA polimerasi, in virtù del ruolo estremamente delicato che ricoprono, sono enzimi spesso complessi e coinvolti in catene di reazioni la cui corretta esecuzione è indispensabile ai fini del mantenimento della vita.
La reazione catalizzata dalle DNA polimerasi porta all'allungamento della catena polinucleotidica di DNA utilizzando un secondo filamento già esistente come stampo e si realizza mediante la formazione di un legame fosfodiesterico tra il gruppo fosfato in posizione 5' del nucleoside da inserire nella catena con il gruppo ossidrile in 3' dell'ultimo nucleoside già presente.
L'energia per la formazione del legame è data dall'idrolisi di due fosfati che vengono rilasciati sotto forma di una molecola di pirofosfato (fig.1).
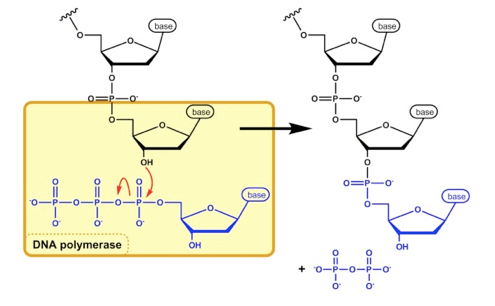
Figura 1: Formazione del legame fosfodiesterico da parte della DNA Polimerasi: Il gruppo ossidrile in 3' reagisce con il gruppo fosfato in 5' . L'idrolisi di due gruppi fosfati genera una molecola di pirofosfato.
In natura si conoscono numerose molecole appartenenti alla famiglia della DNA polimerasi, distinte in:
1) polimerasi eucariotiche;
2) polimerasi procariotiche.
1) DNA polimerasi procariotiche
Nelle cellule procariotiche le DNA polimerasi sono 5, contraddistinte da una numerazione progressiva.
Tra di esse, quella che riveste il ruolo più importante nella sintesi del DNA è la polimerasi III, che attua l'allungamento del filamento sia a carico del filamento lagging che del filamento leading e svolge allo stesso tempo un'attività di "proofreading" ossia di correttore di bozze, esercitata grazie alla funzione di esonucleasi 3'-5' che gli permette, in caso di inserzione del nucleotide errato, di tornare indietro e rimuoverlo per poi inserire nuovamente quello corretto.
La capacità di correggere gli errori è di fondamentale importanza e permette un tasso di errore inferiore a 1 ogni 10.000.000 di nucleotidi.
L'esistenza di un piccolo margine di errore è anch'essa di fondamentale importanza, perché consente l'esistenza di quella piccola variabilità che è il veicolo essenziale per i meccanismi dell'evoluzione.
Le Polimerasi 1,2,4 e 5 hanno invece ruoli minori, essenzialmente legati alla riparazione dei danni, al completamento del lavoro svolto dalla polimerasi III e nella sintesi delle translesioni.
DNA polimerasi III
Come detto, il principale attore della sintesi proteica è l'oloenzima DNA Polimerasi III. Vengono definiti oloenzimi quei complessi multiproteici costituiti da numerose sub-unità di cui una è quella catalitica per la reazione caratteristica dell'enzima e le altre hanno funzione di cofattori, contribuendo a facilitare o amplificare le funzionalità della sub-unità catalitica.
Nel caso della polimerasi III il complesso è formato da due core, che rappresentano le sub-unità catalitiche, unite dal Dimero τ alla cui base si trova il complesso γ (clamp loader) (fig. 2).
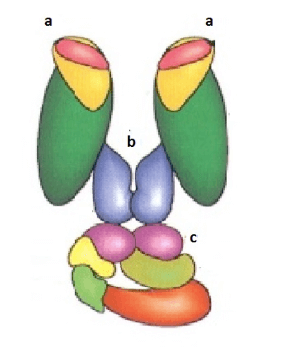
Figura 2: DNA polimerasi III. a) "core" b) Dimero "T" c) complesso "γ".
Prendono parte al sistema anche due unità indipendenti, dette unità β (o sliding clamp), che permettono lo scorrimento del sistema lunga la catena nucleotidica da copiare.
Durante la replicazione del DNA i due core lavorano in contemporanea sui due filamenti permettendo la sintesi simultanea del lagging strand e del leading strand. Il primo step della serie di passaggi che portano alla sintesi del DNA è il legame del complesso γ all'elicasi (che ha prodotto la separazione dei due filamenti stampo della molecola di DNA preesistente); il complesso γ opera quindi il caricamento di due sliding clamp sul filamento stampo e permette l'assemblaggio dei due nuclei catalitici sul DNA.
A questo punto i core iniziano a catalizzare la sintesi progressiva dei vari nucleotidi producendo l'allungamento del filamento.
Poiché la DNA Polimerasi III necessita sempre di uno starter nucleotidico, prima di poter entrare in azione è necessario che una PRIMASI abbia prodotto una breve sequenza di RNA lasciando un gruppo OH 3' libero per la reazione con il gruppo fosfato del primo nucleotide inserito.
La sintesi del filamento veloce (leading strand) determina la formazione di un'ansa lungo il DNA che apre lo spazio alla contemporanea lavorazione, da parte dell'altro core, del filamento lento (lagging strand).
Durante la sintesi di quest'ultimo, il centro catalitico viene riutilizzato al termine di ogni frammento di Okazaki per la sintesi di quello nuovo, mediante un meccanismo per cui il core si stacca dalla clamp ma rimane associato alla forca di replicazione perché connesso al core del filamento veloce; il clamp loader carica altre due sliding clamp in corrispondenza del punto di innesco successivo e il processo riparte.
La processività (ossia la capacità di unire più nucleotidi durante un singolo evento di connessione) in questo enzima è molto alta arrivando a inserire fino a 1000 nucleotidi al secondo.
L'azione della DNA polimerasi III avviene in associazione ad un più ampio complesso di enzimi che prende il nome di Replisoma e che vede coinvolti, oltre all'oloenzima DNA polimerasi III e l'elicasi, anche gli enzimi Girasi, Ribonucleasi, SSB e Ligasi.
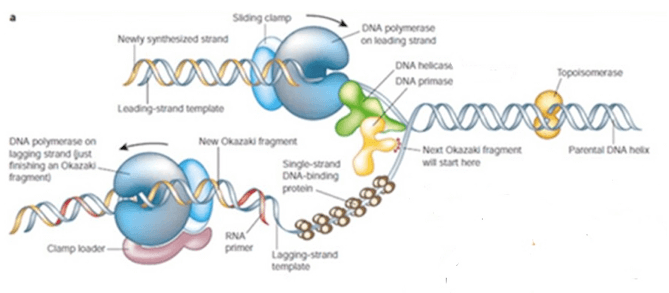
Figura 3: La replicazione del DNA avviene contemporanemente su entrambi i filamenti. I "core" sono associati alle sliding clamp e alle altre componente del Replisoma.
Come detto, anche altri enzimi fanno parte del gruppo delle DNA Polimerasi batteriche.
DNA polimeasi I
Le funzioni associate a questo enzima sono essenzialmente legate alla capacità di rimuovere i PRIMER di RNA, riparare eventuali danni subiti dalle molecole e riempire gli spazi vuoti lungo la sequenza nucleotidica derivati, ad esempio, dalla rimozione dei PRIMER operata dalla polimerasi I stessa.
La struttura della molecola è a singolo filamento, ma con una debole proteolisi può essere scissa in due filamenti di lunghezza differente, che corrispondono alle sequenze con proprietà sintetica (il più lungo, detto filamento di Klenow) o esonucleasica (il più breve). In vitro è utilizzata per innescare la replicazione partendo da un incisione di DNA a filamento singolo secondo il processo definito di spostamento dell'incisione.
DNA polimerasi II
Data la capacità di sintetizzare catene nucleotidiche senza la necessità di uno starter, la DNA polimerasi II è particolarmente adatta a svolgere il ruolo di Gap Filling, cioè di riempimento dei vuoti e di riparazione. Per la bassa processività che la caratterizza essa però non è adatta alla sintesi di lunghe catene
DNA polimerasi IV e V
Queste due molecole abbastanza simili, appartenenti entrambi alla famiglia Y, sono coinvolte essenzialmente nella riparazione del DNA danneggiato in relazione all'attivazione di risposte di tipo SOS per cui la cellula, rilevata la presenza di un danno al materiale genetico, interrompe le attività di replicazione cellulare e attiva meccanismi di riparazione .
DNA polimerasi eucariotiche
Nelle cellule eucariotiche si conoscono 6 differenti tipi di DNA polimerasi, con caratteristiche talvolta peculiari in relazione alla diversa organizzazione interna della cellula eucariotica rispetto a quella procariotica.
Il corrispettivo della DNA polimerasi III nelle cellule eucariotiche è la Polimerasi δ, che è il principale enzima per la sintesi dei filamenti sia lagging che laeading.
Differentemente dalle cellule procariotiche, però , vi è un enzima specializzato nella sintesi dei filamenti lenti, la polimerasi ε.
Ancora, la polimerasi γ è deputata alla replicazione del solo DNA mitocondriale, e quindi non presente nei procarioti.
La polimerasi ɑ è responsabile della sintesi della prima parte del filamento, immediatamente dopo gli starter di RNA.
Le polimerasi β e η sono infine responsabili, rispettivamente, della riparazione dei DNA danneggiati e della sintesi da translesione.
Link correlati:
Quanto è lungo il DNA?
Quali sono gli acidi nucleici?
Qual è la differenza tra DNA e RNA?
Funzioni e struttura dell'apparato di Golgi
Che cos'è l'acido ribonucleico (RNA)?
Che cos'è la reazione a catena della polimerasi o PCR?
Studia con noi