Frammenti di Okazaki
Che cosa sono i frammenti di Okazaki?
Con il termine frammenti di Okazaki vengono comunemente indicate piccole sequenze nucleotidiche prodotte durante la replicazione del “filamento lento” del DNA; prendono il nome dallo scienziato che per primo riuscì a dimostrarne l'esistenza svelando alla comunità scientifica dettagli importanti sul funzionamento delle vie metaboliche necessarie alla replicazione della vita.
La presenze di queste sequenze, dimostrata sia nelle cellule eucariotiche (tipicamente 1000/2000 nucleotidi) che nelle cellule procariotiche (più brevi, poche centinaia di nucleotidi) consentono alle cellule la corretta replicazione del DNA bypassando i limiti intrinsechi degli enzimi coinvolti in questa delicata sequenza di reazioni.
Struttura del DNA
La molecola di DNA è formata da due filamenti complementari costituiti da una sequenza ben precisa di nucleotidi.
I nucleotidi sono molecole composte da uno zucchero pentoso (cioè a cinque atomi di carbonio) a cui è legata una base azotata e un gruppo fosfato.
Sulla molecola di zucchero, che per il DNA è il desossiribosio (fig.1), il carbonio in posizione 1' lega la base azotata, quello in posizione 5' il gruppo fosfato e quello in posizione 3' un gruppo ossidrile.
Il legame tra nucleotidi adiacenti lungo la catena si determina mediante realizzazione di un legame fosfodiesterico tra il gruppo ossidrile in 3' e il gruppo fosfato in 5' cosi che ciascun filamento terminerà con un 3' libero da una estremità e un 5' libero dall'altra, ma in maniera opposta, facendo corrispondere all'estremità 5' di un filamento l'estremità 3' dell'altro (fig.2).
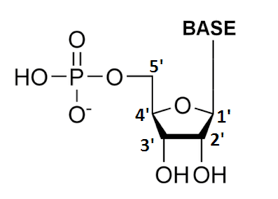
Figura 1: Schema di un nucleotide del DNA: sugli atomi di carbonio 3' e 5' sono situati rispettivamente il gruppo ossidrile e il gruppo fosfato coinvolti nei legami fosfodiesterici lungo la catena
Replicazione del DNA
La replicazione del DNA è di tipo semiconservativo: ciò significa che, per la formazione di una nuova molecola, una molecola già esistente viene divisa e ciascuna delle due metà viene incorporata nella molecola nascente, in cui pertanto solo il 50% sarà neosintetizzato, e la parte preesistente fà da stampo per la sintesi dell'altra, essendo le due complementari.
Il processo di replicazione non inizia in un punto casuale, ma in corrispondenza di precise sequenze che prendono il nome di Origine Della Replicazione; un'enzima, detto elicasi, produce la rottura dei legami esistenti tra nucleotidi complementari dei due filamenti e la separazione degli stessi, generando la cosiddetta Bolla di Replicazione in cui i filamenti separati sono utilizzati come stampo per sintetizzare quelli nuovi.
La DNA Polimerasi, che è l'enzima coinvolto nella sintesi del DNA ha la necessità, per iniziare il processo di sintesi del filamento, di avere già a disposizione un nucleotide con una estremità 3' libera; pertanto il processo inizia con la sintesi di un innesco, detto PRIMER, di RNA operata dall'enzima PRIMASI. L'innesco sarà poi rimosso da enzimi aventi attività esonucleasica.
La sintesi avviene lungo entrambi i filamenti e procede su entrambi nella stessa direzione, pertanto avverrà in direzione 5'- 3' lungo un filamento e in direzione 3'-5' lungo l'altro.
Poiché la DNA Polimerasi, può procedere solo in direzione 5'-3', la formazione dei frammenti di Okazaki è la soluzione adottata dai sistemi biologici per portare a compimento la sintesi di entrambi i filamenti.
Gli esperimenti di Okazaki
Nel 1967 lo scienziato giapponese Reiji Okazaki svelò il meccanismo attraverso cui avviene la sintesi del filamento in direzione 3'- 5'; egli condusse una serie di esperimenti su colture di Escherichia coli che erano in fase attiva di replicazione del DNA, esponendole alla presenza di nucleotidi radioattivi (nello specifico Timina Triziata, cioè Timina contenente Trizio, un isotopo radioattivo dell'idrogeno), per tempi di esposizione crescenti.
L'osservazione fu che, quando le colture erano esposte per tempi brevi alla presenza dell'isotopo radioattivo, questo veniva incorporato esclusivamente in sequenze brevi di DNA, mentre quando l'esposizione era più prolungata esso veniva ritrovato in sequenze molto più lunghe. Le sequenze brevi erano composte da circa 100/200 nucleotidi.
Esperimenti analoghi, svolti poi su cellule eucariotiche, dimostrarono che il fenomeno si ripeteva anche in esse ma generando sequenze comprese tra 1000 e 2000 nucleotidi.
Conclusioni
La conclusione a cui Okazaki arrivò è che i due filamenti vengono sintetizzati in maniera differente: il filamento disposto in posizione 5'- 3', cioè la direzione seguita dalla polimerasi e che prende il nome di “filamento leader” o “filamento veloce” (in inglese “ leading strand”) viene sintetizzato tutto d'un fiato, a partire da un unico innesco; il filamento complementare (“filamento lento” o “lagging strand”), disposto in direzione 3'- 5', viene invece sintetizzato, per così dire, “a pezzi”: l'innesco viene generato in posizione un po' avanzata rispetto all'origine della replicazione e la polimerizzazione avviene in senso contrario al senso di allungamento del filamento, viaggiando quindi in direzione 5'- 3' verso l'origine della replicazione e non allontanandosi da essa, producendo solo un frammento di DNA.
Il processo si ripete in continuazione procedendo così per piccoli frammenti, ognuno separato dai successivi dalla presenza di un ulteriore PRIMER di RNA. Tali frammenti sono detti, appunto, frammenti di Okazaki (fig.2).
Alla fine i PRIMER vengono rimossi dall'enzima RNAsi e sostituiti da DNA ad opera dell'enzima DNA Polimerasi I.
L'azione dell'enzima DNA ligasi permette la giunzione di tutti i frammenti generati mediante formazione dei legami fosfodiesterici tra i gruppi OH delle estremità 3' e i gruppi fosfato sulle estremità 5' dei frammenti adiacenti.
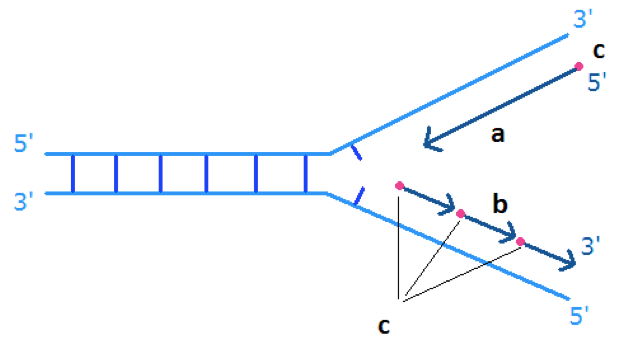
Figura 2: Replicazione del DNA: a) filamento veloce; b) Filamento lento con frammenti di Okazaki; c) Primer d'innesco per la DNA Polimerasi.
Sequenze telomeriche
Una importante conseguenza del meccanismo di replicazione del filamento 3'- 5' mediante sintesi dei frammenti di Okazaki è la perdita dell'informazione contenuta alle estremità dei filamenti.
Infatti, quando la replicazione giunge all'estremità del filamento, l'ultimo tratto non viene replicato per l'impossibilità di generare il PRIMER e innescare la DNA Polimerasi.
Per ovviare all'effetto deleterio della perdita di informazione tra generazioni successive al margine dei filamenti sono presenti sequenze ripetute non codificanti, il cui progressivo accorciamento non produce effetti evidenti dal punto di vista fenotipico, anche se potrebbero essere probabilmente collegati ai
Link correlati:
Quanto è lungo il DNA?
Quali sono gli acidi nucleici?
Che cos'è la reazione a catena della polimerasi o PCR?
Studia con noi