Ricombinazione omologa
Che cos'è la ricombinazione omologa?
La ricombinazione omologa è un fenomeno che consiste nello scambio di DNA tra porzioni di filamenti omologhi.
Può avvenire mediante il passaggio di sequenze geniche tra cromatidi fratelli oppure tra cromosomi omologhi e svolge due funzioni essenziali: la ricombinazione omologa è il principale meccanismo di riparazione del DNA danneggiato e rappresenta la via attraverso la quale si originano nuove combinazioni geniche o alleliche durante la meiosi (crossing over).
Meccanismo della ricombinazione omologa
Per illustrare il meccanismo della ricombinazione omologa prendiamo ad esempio un tipico caso in cui questa avviene: la rottura di un doppio filamento di DNA.
In casi come questo, interviene innanzitutto un complesso multiproteico detto MRN, formato dalla proteina MRE11; la proteina RAD50 e la proteina NBS1 (Nibrina).
RAD 50 è una proteina formata da un dimero circolare, che lega il doppio filamento di DNA danneggiato avvolgendolo nel punto di rottura e recluta un dimero di MRE11 e una subunità proteica di NBS1.
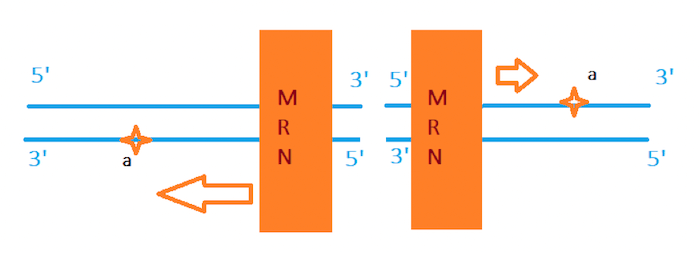
Il complesso MRN riconosce il sito di rottura, vi si lega e scorre sul filamento allontanandosi per circa 100 basi, prima di effettuare il taglio (a) sui filamenti in cui la rottura è all'estemità 5'.
Una volta riconosciuto il sito di rottura ed avviato il legame, MRN si allontana dal punto di rottura di circa 100 basi ed effettua un taglio sul filamento di DNA che presenta l'estremità 5' rivolta verso il punto di rottura.
A questo punto, il complesso inizia a muoversi verso il punto di rottura rimuovendo tutti i nucleotidi che incontra. Tale azione è consentita dalla capacità di MRE11, che possiede attività endonucleasica (per tagliare il filamento) ed esonucleasica (per rimuovere i nucleotidi in sequenza durante l'avanzamento).
Contemporaneamente, un altro enzima esonulceasi (EXO1), reclutato da MRN, taglia il filamento in direzione 3'. Così facendo, si genera un ampia porzione di filamento distrutta.
Questo processo avviene in contemporanea su entrambi i tronconi del doppio filamento danneggiato.
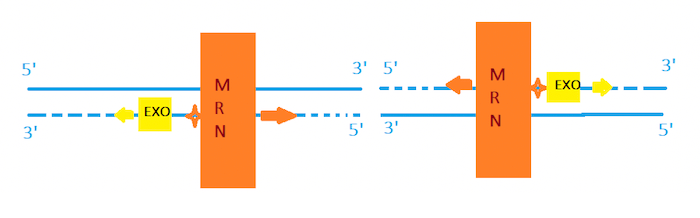
MRN e EXO agiscono come esonucleasi e idrolizzano una porzione abbondante dei filamenti.
Si generano a questo punto due lunghe porzioni di DNA a singolo filamento, che vengono avvolte da una proteina con funzione protettiva (RPA), che viene sostituita da un'altra proteina, RAD51.
Il singolo filamento di DNA che termina con l'estremità 3' libera, ricoperto dalla proteina RAD51, è in grado di invadere un molecola a doppio filamento di DNA e scalzare il filamento ad esso completare in un processo noto come "strand invasion", legandosi al filamento complementare e creando una struttura detta ansa D (o D-Loop).
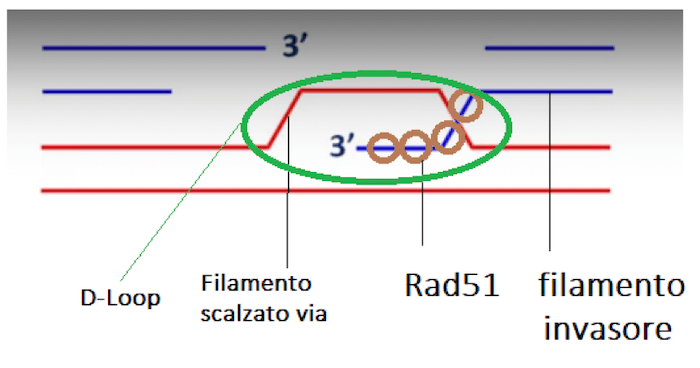
La molecola da riparare presenta due porzioni a singolo filamento, avvolte da Rad51. Una di queste invade una molecola di un cromosoma omologo e scalza via il suo filamento corrispondente, che ripiega formando l'ansa D.
A questo punto la molecola ibrida così formata può andare incontro a due possibili evoluzioni:
- Syntesis-dependent strand annealing (SDSA)
- Holliday Junction
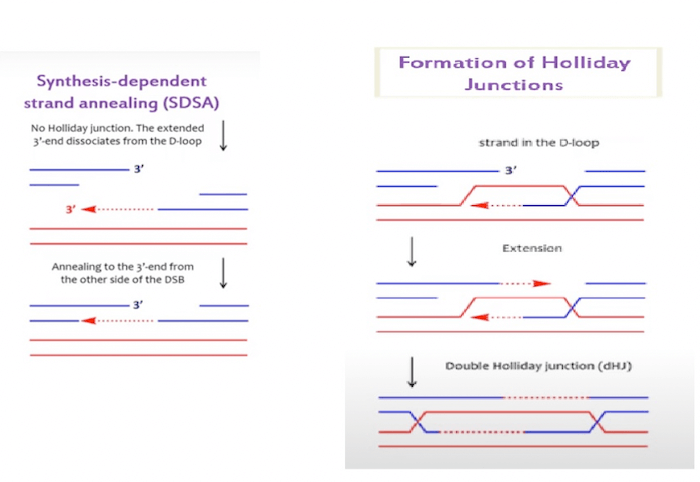
L'invasione del filamento "strand invasion" può risolversi in SDSA oppure si può generare una giunzione di Holliday.
SDSA
In caso di annealing dipendente dalla sintesi di DNA, interviene una polimerasi che lega l'estremità 3' del filamento invasore e, utilizzando la porzione libera del filamento complementare come stampo, allunga il filamento interrotto in direzione 3'.
L'Ansa D scorre verso l'estremità 3' e l'allungamento continua fino a che il filamento non è sufficientemente lungo da ricongiungersi con la sua porzione restante, nel tratto in cui si era interrotto.
L'invasione quindi si risolve e il filamento neosintetizzato fa da stampo per la sintesi della porzione mancante del filamento complementare. Non si ha crossing over ma Gene Conversion (vedi figura precedente).
Il crossing over, infatti, prevede che il prodotto finale sia formato da filamenti formati dalla parte iniziale di uno dei filamenti di partenza e la parte finale di quello ad esso omologo: in poche parole bisogna immaginare che i due filamenti omologhi vengano tagliati in un punto e i tronconi generati vengano interscambiati.
Nel caso della Gene Conversion, invece, le porzioni di DNA precedente e successiva al punto di rottura non hanno subito variazione, mentre la porzione interessata dalla rottura ha acquisito l'informazione genetica della molecola omologa.
Vi è quindi un piccolissimo scambio di informazione genetica, che coinvolge solo la molecola oggetto della riparazione, mentre l'altra rimane assolutamente immutata.
Holliday Junction
Si ha la formazione della giunzione di Holliday se l'estremità 3' libera del filamento complementare a quello che ha invaso l'altra molecola di DNA forma legami con la porzione ad esso complementare del filamento che ha formato l'ansa D.
L'ibridazione che si genera fa da primer per una polimerasi che sintetizza nuovo DNA andando a chiudere il filamento. L'ansa D scorre verso l'estremità 3' e il filamento invasore si ricongiunge al tratto mancante.
La struttura che si forma sarà caratterizzata da due giunzioni di Holliday, che potranno risolversi in 3 modi: Mediante dissoluzione, mediante risoluzione con crossing over o mediante risoluzione senza crossing over.
Dissoluzione: l'ansa D, che si era allargata durante la sintesi del DNA, restringe nuovamente fino a che i due filamenti si ritrovano uniti come in una maglia di catena.
A questo punto un enzima specifico taglia uno dei due filamenti consentendo all'altro di sfilarsi, e poi ricuce il taglio. Anche in questo non si ha crossing over (figura seguente).
Risoluzione con o senza crossing over
Per capire questo meccanismo, dobbiamo ridisegnare la giunzione di Holliday in proiezione planare, come in figura.
La giunzione si risolve mediante un taglio nel punto di incrocio dei filamenti, che può avvenire lungo il piano orizzontale o il piano verticale.
Il taglio verticale produce due cromosomi ricombinanti e da luogo a crossing over, mentre il taglio lungo il piano orizzontale non da luogo a crossing over e le uniche porzioni dei cromosomi in cui c'è scambio genico sono quelle a cavallo della rottura iniziale.
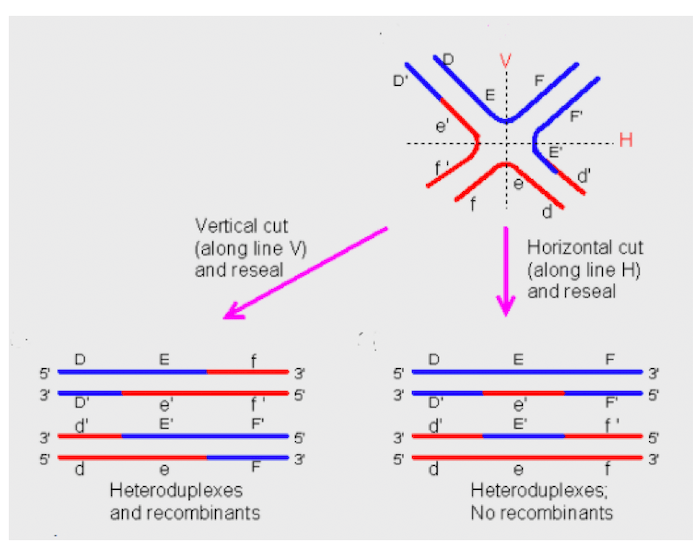
Un taglio lungo il piano verticale da origine ad un crossing over.
Nel caso della doppia giunzione di Holliday, come nell'esempio fino a qui descritto, si ottiene un evento di crossing over solo se i tagli avvengono su piani differenti.
Qualora i tagli avvenissero sullo stesso piano, infatti, si otterrebbero cromosomi non ricombinanti (fatta eccezione, come sempre, per la porzione che comprende la rottura).
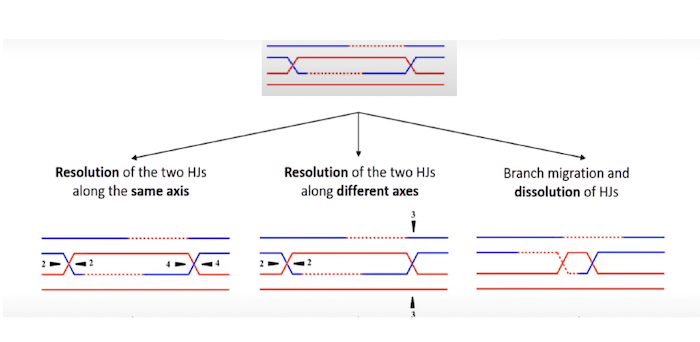
Le tre vie di uscita dalla doppia giunzione di Holliday: Risoluzione senza crossing over (sx); risoluzione con crossing over (centro) e Dissoluzione senza crossing over (dx).
Conclusioni
Il meccanismo di ricombinazione omologa, in definitiva, è quel processo attraverso cui si ottiene lo scambio di materiale genetico tra molecole di DNA omologhe oppure identiche.
Nella maggior parte dei casi il DNA utilizzato come aiuto alla riparazione è quello di un cromosoma omologo. Questo è tanto più vero quando si osservi la ricombinazione omologa che avviene in Profase I della meiosi.
Attraverso questi eventi, che sono determinanti per il riassortimento dei geni nei cromosomi, gli eucarioti si assicurano di massimizzare gli effetti positivi della riproduzione sessuata e riparare i danni subiti dai cromosomi, specialmente durante le fasi di replicazione del DNA.
Link correlati:
Quanto è lungo il DNA?
Che cos'è il sequenziamento del DNA?
Che cos'è la reazione a catena della polimerasi o PCR?
Studia con noi