Crossing-over
Che cos'è il crossing-over?
Il crossing-over è un evento di ricombinazione genica che avviene durante la Profase I della meiosi e che coinvolge sequenze omologhe di DNA su cromatidi non fratelli di cromosomi omologhi. Rappresenta pertanto uno dei principali fattori di determinazione della variabilità genetica, che è a sua volta un elemento di fondamentale importanza per il mantenimento dell'integrità delle specie, nonché caratteristica indispensabile per il processo evolutivo.
La scoperta del crossing-over ha permesso, tra l'altro, di dare risposta all'incompleta corrispondenza delle previsioni dettate dalle leggi di Mendel rispetto all'effettivo assortimento dei geni nella progenie. Ogni evento di crossing-over determina, per ogni cromosoma, la formazione di 50% di gameti ricombinanti, ossia gameti in cui la sequenza genica contenuta nel cromosoma è diversa da quella del cromosoma di partenza.
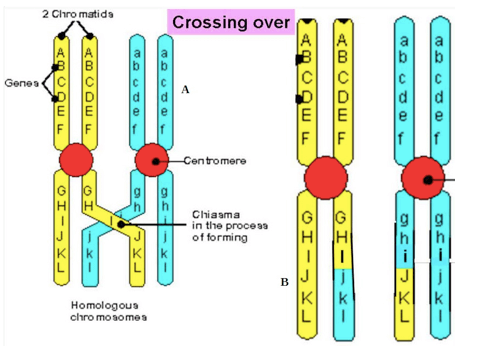
Figura 1: Crossing-over: dopo la formazione del chiasma ("A") si ha la ricombinazione di una porzione dei cromatidi ("B").
Importanza del crossing-over
Sebbene l'effetto più rappresentativo associato al crossing-over sia l'aumento esponenziale del differenziamento genetico all'interno di una popolazione, forse quello più importante è la capacità di stabilizzare un altro processo, quello di segregazione dei cromosomi. Infatti, in presenza di mutazioni che coinvolgono geni determinanti per la corretta esecuzione del crossing-over si determina un fallimento della segregazione dei cromosomi in percentuali superiori al 90%. È probabile che la formazione del "chiasma" permetta la corretta associazione dei cromosomi al fuso e la corretta separazione degli stessi al termine della Profase I.
La Profase I della meiosi
La meiosi è quel processo di divisione cellulare che porta alla formazione di 4 cellule aploidi a partire da una cellula madre diploide. Si divide in due fasi: durante la fase 1 si ha la separazione dei cromosomi omologhi, durante la fase 2 si ha invece la separazione dei cromatidi fratelli.
La Profase 1 è caratterizzata da 5 momenti:
- Il Leptotene, in cui i cromosomi compaiono sotto forma di sottili filamenti
- Lo Zigotene, in cui i cromosomi omologhi si congiungono a formare le sinapsi
- Il Pachitene, in cui i cromosomi si accorciano e si inspessiscono e le coppie di cromosomi vanno a formare i "bivalenti"
- Il Diplotene: i cromosomi si sdoppiano, appaiono le coppie di cromatidi fratelli e si forma la "tetrade", in cui i quattro cromatidi sono congiunti in un'unica struttura. In questa fase avviene il crossing-over.
- In seguito al Diplotene si ha la scomparsa del nucleo e la formazione del fuso.
Caratteristiche del crossing-over
Come detto, il crossing-over avviene durante la Profase I della meiosi precisamente dopo la formazione delle tetradi che consente lo stretto contatto tra i cromatidi non fratelli. A questo punto avviene la rottura dei filamenti e la formazione del "chiasma", ossia una giunzione tra specifiche regioni di cromatidi non fratelli che determina la momentanea associazione dei due cromosomi omologhi in un unico complesso.
La formazione del chiasma è evidente e osservabile anche al microscopio, e permette lo scambio di una porzione del filamento tra i due cromosomi generando un cromatidio ricombinante.
Durante la profase I tutte le coppie di cromosomi omologhi formano almeno un chiasma; non è raro che se ne formi più di uno. Ogni filamento può ricombinare con ciascuno dei filamenti del cromosoma omologo, anche con entrambi in punti differenti. Si possono pertanto verificare crossing-over singoli, doppi, tripli o quadrupli.
I punti in cui avviene il crossing-over non sono casuali e lungo le sequenze nucleotidiche vi sono regioni a più alta frequenza di ricombinazione e regioni invece che rappresentano dei blocchi altamente conservati, dove gli eventi di ricombinazione sono assai rari, composti da alcune decine di migliaia di coppie di basi.
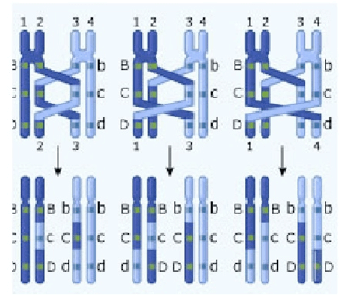
Figura 2: Crossing-over multipli.
Giunzione di Holliday
Una maggiore comprensione del meccanismo di funzionamento del crossing-over ci viene dal modello descritto da Holliday nel 1964 per descrivere la ricombinazione omologa nei batteri.
Secondo questo modello, dopo la rottura dei filamenti del DNA si ha l'invasione, da parte dei filamenti liberi, della sequenza omologa nei filamenti intatti e la formazione di una struttura a croce, detta giunzione di Holliday. Una volta formatasi questa giunzione, attraverso il progressivo appaiamento delle basi complementari, la giunzione si sposta seguendo la direzione 3' – 5'. Il processo termina col taglio dei filamenti in corrispondenza del chiasma e la separazione dei due cromosomi.
Proteine coinvolte nel processo del crossing-over
Per il corretto svolgimento del processo del crossing-over intervengono alcune proteine specifiche:
- Spo11: si tratta della proteina che determina la formazione delle rotture dei filamento di DNA. Entra in funzione nelle primissime fasi dopo l'accoppiamento dei cromosomi omologhi
- MRX: un complesso multiproteico che attacca i filamenti appena aperti dalla proteina Spo11 e determina la degradazione delle estremità in direzione 5' – 3' permettendo l'invasione del filamento.
- DMC1: analogo della RecA batterica, questa proteina è responsabile dell'appaiamento delle basi complementari dei filamenti provenienti da cromosomi differenti e rende possibile l'appaiamento dei filamenti anche in presenza di sequenze non del tutto complementari (formazione dell'eteroduplex).
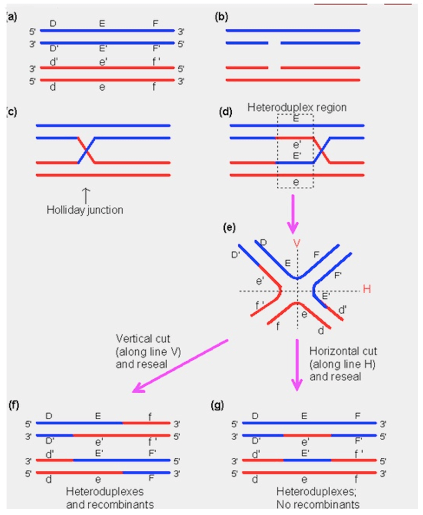
Figura 3: Dettagli del crossing-over: con la formazione del chiasma si ha la creazione della giunzione di Holliday, lo scorrimento del chiasma con formazione della regione eteroduplex e successivamente il taglio nel punto di giunzione. Solo se il taglio avviene lungo l'asse verticale (e) si ha crossing-over (f) , diversamente si ha il ripristino quasi integrale dei cromatidi originali fatta eccezione per una piccola regione intermedia (g).
Crossing-over e mappatura del genoma
Il celebre scienziato Thomas Hunt Morgan, genetista, embriologo ed evoluzionista americano, intuì che geni posizionati su loci più distanti ricombinano e si separano con maggiore frequenza di geni posizionati in loci adiacenti, che invece tendono a rimanere associati e segregare insieme, dividendosi con minore frequenza. In base a questa intuizione fu proposto il primo sistema di mappatura del genoma, prendendo la frequenza di ricombinazione come riferimento per posizionare, relativamente gli uni agli altri, in vari geni lungo il cromosoma.
L'unità di misura proposta fu il CentiMorgan che rappresentava la distanza genica dell'1%, ossia una ricombinazione ogni 100 eventi di crossing-over. L'utilizzo della frequenza di ricombinazione come indice della distanza genica è soggetto ad una diminuzione dell'accuratezza al crescere della distanza tra i geni, poiché risente degli effetti del crossing-over doppio.
In caso di doppio evento di ricombinazione infatti, il risultato è il ripristino della iniziale sequenza genica ad eccezione di una piccola regione a livello del gene intermedio. La massima frequenza di ricombinazione è del 50%, per cui la massima distanza possibile esprimibile in CentiMorgan è 50. Geni assolutamente associati hanno una distanza pari a 0, cioè non segregano mai indipendentemente.
Un'altra ragione che determina la scarsa accuratezza di questo metodo è la differenza nella frequenza delle ricombinazioni (RF) tra le zone definite HotSpot per la ricombinazione e le zone a RF più bassa. In generale, le zone HotSpot sono caratterizzate da una conformazione della cromatina detta Eucromatina, meno compatta e quindi più facilmente soggetta all'azione delle proteine responsabili del crossing-over. Le zone a bassa frequenza di ricombinazione hanno invece cromatina molto più condensata (Eterocromatina) e sono per tanto più protette.
Link correlati:
Quali possono essere gli effetti fenotipici delle mutazioni geniche?
Che cosa sono le mutazioni genetiche?
Che cos'è la clonazione?
Che cosa è la deriva genetica?
Studia con noi