Introni ed esoni
Che cosa sono introni ed esoni?
Introni ed esoni sono due tipologie di sequenze nucleotidiche del DNA. Mentre le sequenze codificate dagli esoni rappresentano il codice espresso dagli mRNA maturi, le sequenze di RNA corrispondenti agli introni sono rimosse dall'RNA in fase di maturazione, non apportando quindi nessun contributo alla sintesi proteica.
La funzione più probabile degli introni è di tipo regolatorio, essendo responsabili della regolazione dell'espressione genica secondo modalità differenti. Alcuni geni sono però contenuti come parte di altri geni di dimensioni maggiori; in una condizione di questo tipo, sequenze non codificanti (introni) del primo gene possono rappresentare invece sequenze codificanti (esoni) del secondo.
La presenza di introni nel DNA è riscontrata negli eucarioti e negli archeobatteri, ma non negli eubatteri, suggerendo, secondo alcuni, la derivazione degli eucarioti a partire dagli proprio dagli Archea.
Dal DNA alle proteine
Il meccanismo che permette di trasferire l'informazione contenuta nei geni in modo da tradurla in sequenze di amminoacidi nella costruzione delle proteine segue tre passaggi fondamentali:
- la trascrizione, in cui il codice del filamento di DNA è usato come stampo per produrre un filamento complementare di RNA messaggero (mRNA);
- la maturazione del mRNA, in cui determinate sequenze di nucleotidi vengono eliminate e il filamento riassemblato solo con le sequenze sopravvissute (splicing);
- la traduzione, che avviene grazie all'intervento dei ribosomi e dell'RNA transfert (tRNA) che determina la corrispondenza tra l'mRNA maturo e la sequenza amminoacidica delle proteine di sintesi.
La rimozione degli introni avviene nella seconda fase, ossia quella della maturazione dell'RNA.
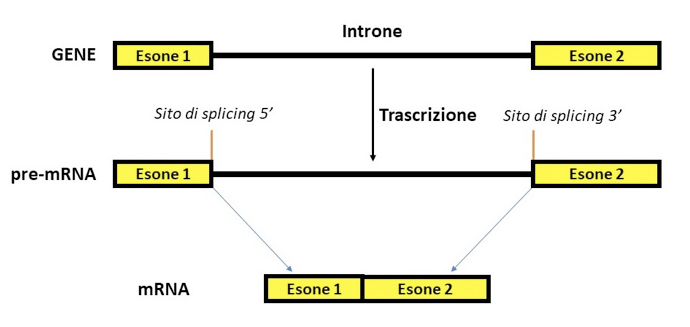
Figura 1: Gli esoni (in giallo) sono la componente del trascritto primario (pre-mRNA) che rimane nel trascritto maturo di mRNA.
Rimozione degli introni nella fase di splicing
La maturazione dell'mRNA avviene grazie all'intervento di specifiche macromolecole, dette spliceososomi, la cui composizione è data dall'assemblaggio di cinque piccole sequenze di RNA ribosomiale che interagiscono in vario modo con oltre 150 proteine andando a formare dei complessi detti ribonucleoproteine nucleari o snRNP. Tali complessi enzimatici sono in grado di riconoscere le sequenze di inizio e fine degli introni nella sequenza nucleotidica del mRNA.
Le estremità degli introni sono caratterizzate da sequenze ripetute di GU all'estremità 5' e AG all'estremità 3', che rappresentano il segnale di attacco per la ribonucleoproteina. Una volta rimossi gli introni, le porzioni gli esoni adiacenti vengono saldati tra loro per la ricostituzione di un unico filamento.
Le dinamiche di interazione tra i vari componenti delle snRNP sono mutevoli e mostrano variazioni significative durante il processo di rimozione degli introni.
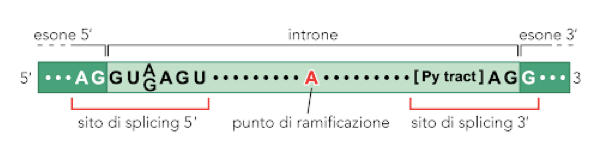
Figura 2: le estremità 3' e 5' degli introni presentano sempre, rispettivamente, le sequenze AG e GU che ne consentono il riconoscimento.
La rimozione degli introni deve avvenire necessariamente in modo perfetto. La rimozione di un nucleotide in più o in meno comporta generalmente la produzione di una proteina totalmente inefficace. Questo perché, essendo il codice contenuto negli acidi nucleici formato da triplette codificanti per un singolo amminoacido, la rimozione o l'aggiunta di una base comporta lo slittamento di tutta la sequenza a valle dell'errore e quindi il cambiamento totale del significato della sequenza restante.
Introni e regolazione dell'espressione genica
Gli introni sono una componente essenziale della complessa regolazione dell'espressione genica negli organismi più evoluti. Alcuni di essi infatti contengono regioni di DNA che funzionano da promotori ed enhancer dell'espressione dei geni a valle, cioè promuovono una maggiore attività di trascrizione a carico di questi ultimi, oppure costituiscono siti di attacco per i loro repressori. Ma la funzione più interessante è quella collegata allo splicing alternativo e quindi nella regolazione post- trascrizionale dell'espressione genica.
Il processo di splicing alternativo, che regola l'espressione del 90% di geni nell'uomo, consiste in un processo attraverso il quale lo stesso trascritto primario di mRNA immaturo viene modellato in maniera differente, in fase di maturazione, attraverso la rimozione di parte degli esoni oltre che degli introni.
Lo splicing alternativo è possibile grazie alla presenza di siti di inizio e fine sugli introni che hanno maggiore o minore affinità con i siti di riconoscimento degli spliceosomi, così in risposta a varie condizioni intranucleari si può verificare una rimozione dei soli introni o la rimozione di esoni contenuti a cavallo di uno o più sequenze introniche.
Il risultato è la produzione di trascritti maturi che condurranno alla produzione di proteine simili, talvolta coinvolte nei medesimi processi metabolici, ma con funzionalità differenti e in alcuni casi opposte.
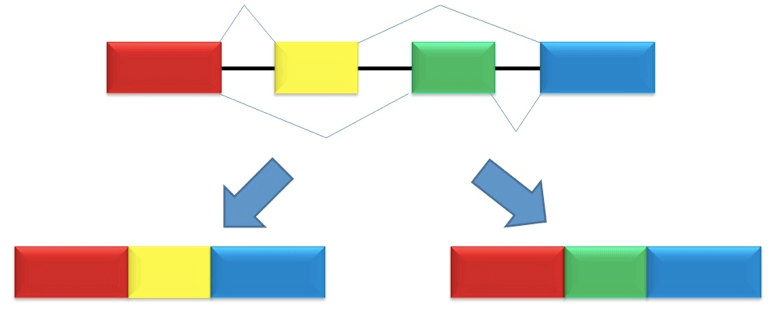
Figura 3: Attraverso il meccanismo dello splicing alternativo il medesimo trascritto primario può portare alla produzione di mRNA differenti mediante la rimozione selettiva degli esoni alternativi.
Un altro meccanismo connesso allo splicing alternativo è il riconoscimento di siti di taglio differenti, ricadenti nello spazio occupato dalle sequenze esoniche, con conseguente modifica della porzione codificante del trascritto, oppure il mantenimento di sequenze introniche che non vengono rimosse.
Gli esoni di un gene che sono presenti in ogni trascritto che da questo deriva sono detti esoni costitutivi, mentre quelli che possono selettivamente essere mantenuti o rimossi sono detti esoni alternativi. Le varie proteine che si generano attraverso lo splicing alternativo di una gene sono definite isoforme di quel gene.
Esoni non codificanti ed esonizzazione degli introni
Abbiamo visto come la corrispondenza tra un trascritto primario di RNA messaggero e le proteine codificate dal gene sia qualcosa di plastico e soggetto a profonda regolazione.
La plasticità di questi meccanismi è aumentata dal fatto che non tutti gli esoni sono in realtà codificanti, ma alcune sequenze in essi contenute, e quindi presenti negli mRNA maturi, non sono in grado di codificare per sequenze di amminoacidi ma costituiscono le porzioni UTR (untraslated region) alle estremità del trascritto.
Inoltre, alcune mutazioni possono alterare i siti di attacco delle ribonucleoproteine e rendere le sequenze introniche non più escisse dal trascritto maturo, trasformandole pertanto in esoni.
Link correlati:
Spiegazione della seconda legge di Mendel
Spiegazione della prima legge di Mendel (legge della dominanza)
Che cos'è l'ontogenesi?
Che cosa si intende per dominanza incompleta?
Studia con noi